主成分分析
通常高通量数据中含有很多变量,主成分分析是一种数据降维方法,利用正交变换把原始的可能相关的变量转换为一组正交新变量, 提取数据中重要的特征,去除不重要的特征(噪声)。方差越大,表示的特征信息越多,的选择方差最大的方向,去除方差较小的方向。
比如微生物组的16S rRNA测序数据,通常每个样品会含有多个OTU(假设有500个)。为了根据OTU丰度对不同分组的样本进行分类,每个OTU的丰度差异都可以在一定程度上反应样品之间的差异,但是不同的OTU之间可能存在着一定的相关性,可能会造成信息的冗余。PCA就是在保持原有变量所包含信息的前提下,减少变量个数进行分析。
PCA原理 - 奇异值分解
假设有一组包含个变量的数据,含有个样本,构成了一个的矩阵。用图形表示,是指在空间内的个点,当的每一行都减去该行的均值后(数据中心化),个点通常聚集在一条线或者平面周围(或其他的低维子空间)。
以含两个变量的个点为例说明PCA的步骤, 维度为。的每一行减去该行的均值,使中心化,每一行的均值为0。如图所示,这个点分布在一条直线周围。由图中也可以直观的看到,主成分方向还表示原始数据在该方向上投影方差最大(分散),与之垂直的投影距离最小。
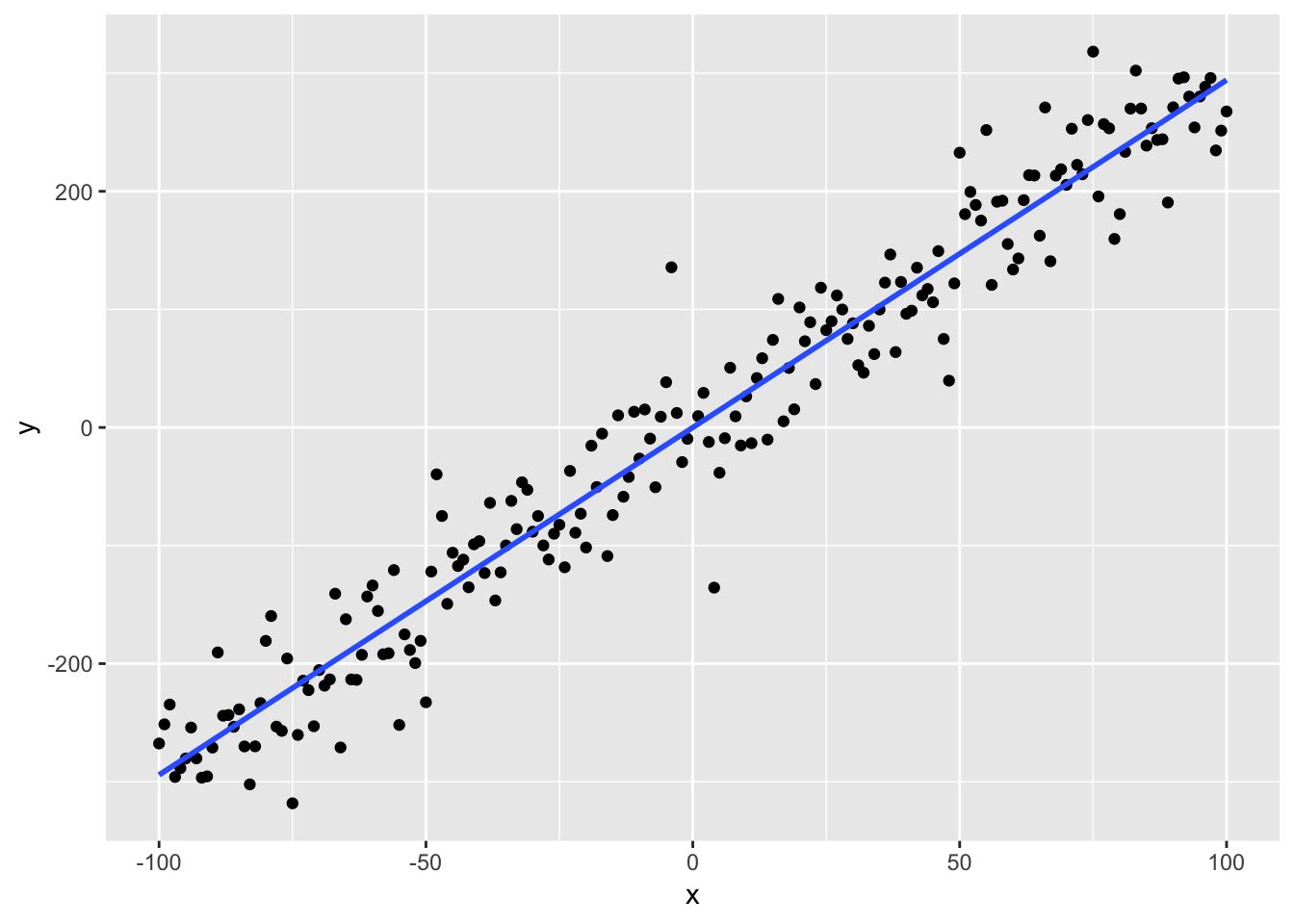
构成一个的矩阵,其中和是行向量的方差,而和即是两行的协方差。因此,协方差矩阵可表示为
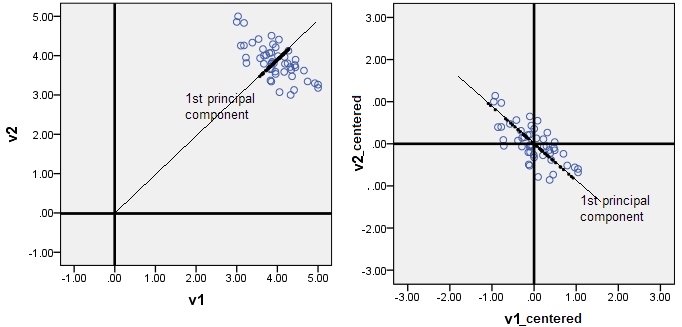
中心化的原因:保证第一个主分量是方差最大的方向,如果不中心化,那么第一主成分的方向受均值的影响而有一定的倾向性,如下图。
求协方差矩阵的特征值和。根据矩阵奇异值分解公式,中心化后的可表示为
假设中心化后为(各行和为0)
特征值为57和3,那么可表示为
对应于散点图变化最大的方向(直线),垂直于,表征垂直于直线方向上的小幅摆动。
所以,对于多维变量,把原始数据转换到向量构成的空间内,特征值越大表征在该向量的方向上数据变异越大。通常选择变异量较大的前几个变量用于表征数据。
PCA数学推导
以最大投影方差法推导,原始数据的中心点为:
原始数据向投影向量投影之后的方差为:
根据梯度优化原则(拉格朗日乘子):
因此,对于多维数据,协方差矩阵的特征值即为方差最大方向,从公式中也可看出PCA分析需要预先对数据中心化。
相关系数矩阵
协方差矩阵是没有消除量纲的表示变量之间关系。当变量的单位对结果有影响的时候。通常选择消除量纲的相关矩阵进行PCA分析。
协方差:
相关系数:
所以标准化后的协方差就是相关系数。和标准化
主成分分析R代码实现
以mtcars数据为例,PCA是针对数值数据,所以我们删除mtcars中的分类变量vs和am。
library(dplyr)
# 首先中心化,因为各变量量纲不同scale = TRUE,
# 主成分是原始变量的线性组合,$rotation表征了线性组合系数,x
# 表示新的坐标值
mtcars_pca <- prcomp(select(mtcars, -c("vs", "am")),
center = TRUE, scale = TRUE)
summary(mtcars_pca)## Importance of components:
## PC1 PC2 PC3 PC4 PC5 PC6
## Standard deviation 2.3782 1.4429 0.71008 0.51481 0.42797 0.35184
## Proportion of Variance 0.6284 0.2313 0.05602 0.02945 0.02035 0.01375
## Cumulative Proportion 0.6284 0.8598 0.91581 0.94525 0.96560 0.97936
## PC7 PC8 PC9
## Standard deviation 0.32413 0.2419 0.14896
## Proportion of Variance 0.01167 0.0065 0.00247
## Cumulative Proportion 0.99103 0.9975 1.00000共9个主成分32个线性相关变量重新组合成9个正交变量,第一个主成分PC1解释了总体数据63%的特征,PC2解释了23%的总体特征,PC1和PC2解释了86%的数据特征,因此仅仅通过前两个主成分就能基本确定样本的位置。
PCA结果可视化
biplot显示样本在新的坐标下的位置,同时显示原始的变量值(根据rotation在相应的主成分标出原始值),向量箭头起始于中心点。
library(ggbiplot)
ggbiplot(mtcars_pca, labels=rownames(mtcars))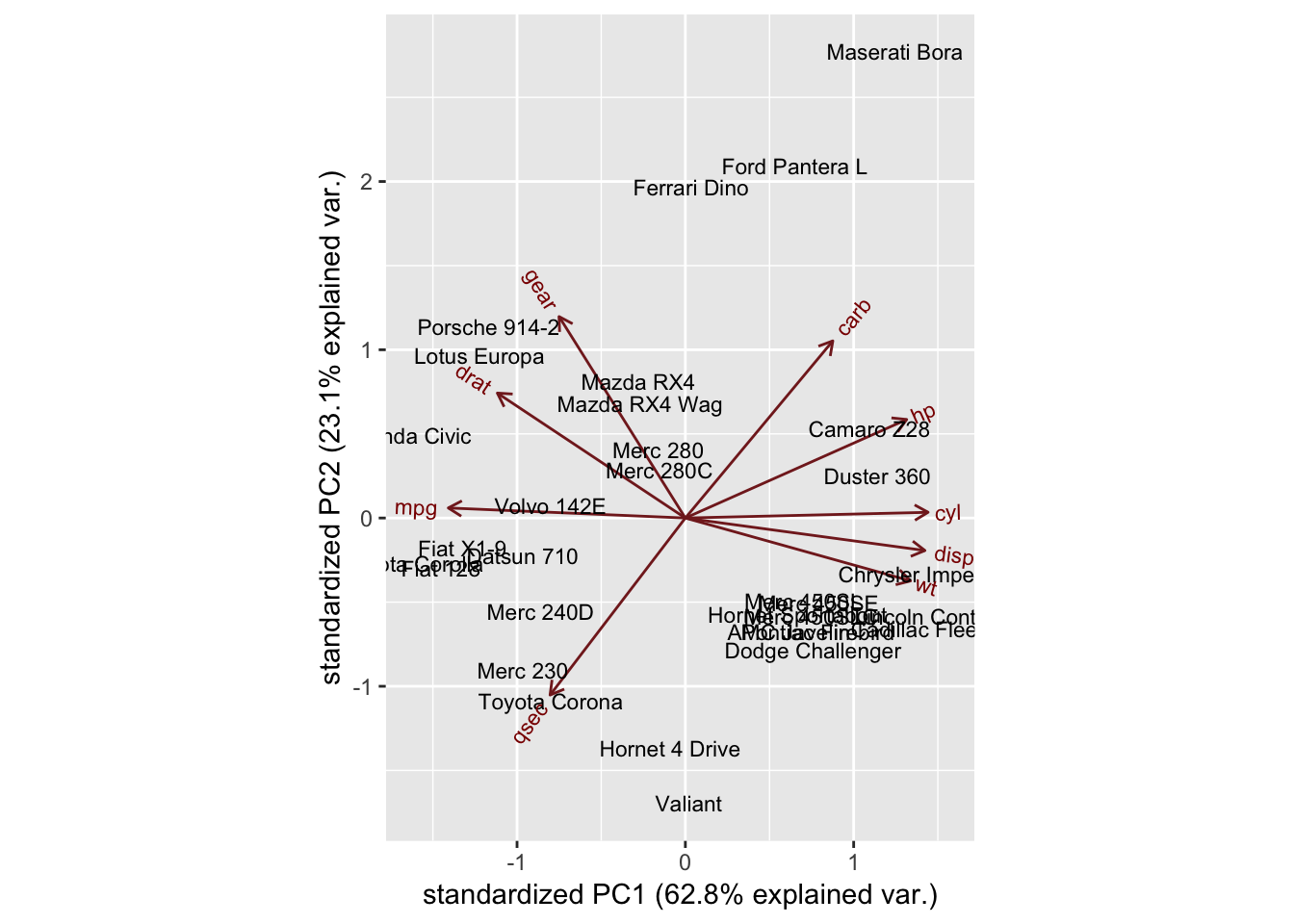
从图中可以看出,变量cyl、disp、wt、hp值较高的样本倾向于位于图中右侧。显示这些变量与哪些样品有关。
结果解读
对车进行分类,观察车出产地情况
mtcars_country <- c(rep("Japan", 3), rep("US",4), rep("Europe", 7),rep("US",3), "Europe", rep("Japan", 3), rep("US",4), rep("Europe", 3), "US", rep("Europe", 3))
ggbiplot(mtcars_pca,ellipse=TRUE, labels=rownames(mtcars), groups=mtcars_country)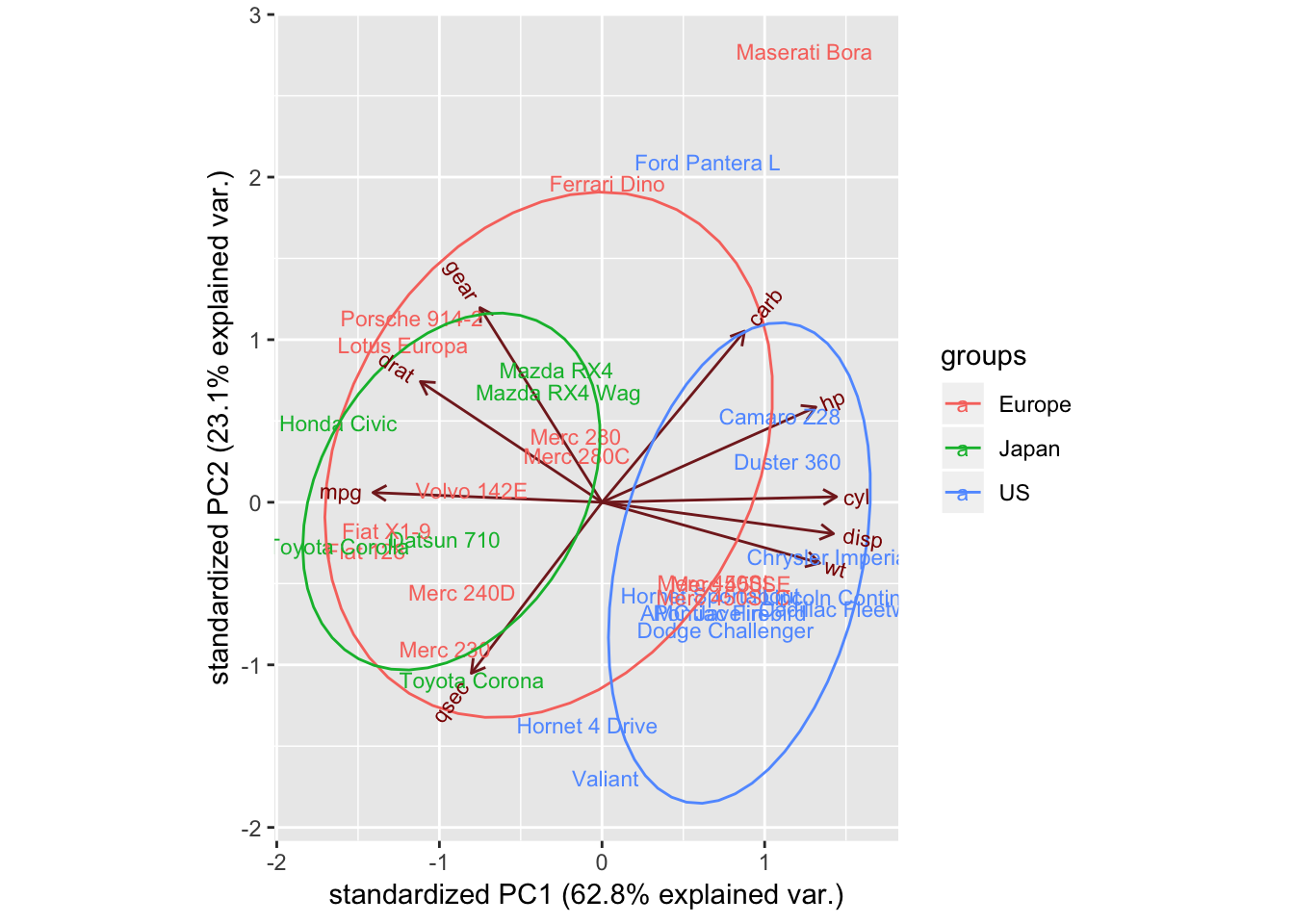
美国产汽车聚集到右侧,且cyl,disp,wt较大;日本产汽车mpg较大,欧洲产汽车位于中间且相对分散一些。
第三和四主成分情况
ggbiplot(mtcars_pca,ellipse=TRUE,choices=c(3,4), labels=rownames(mtcars), groups=mtcars_country)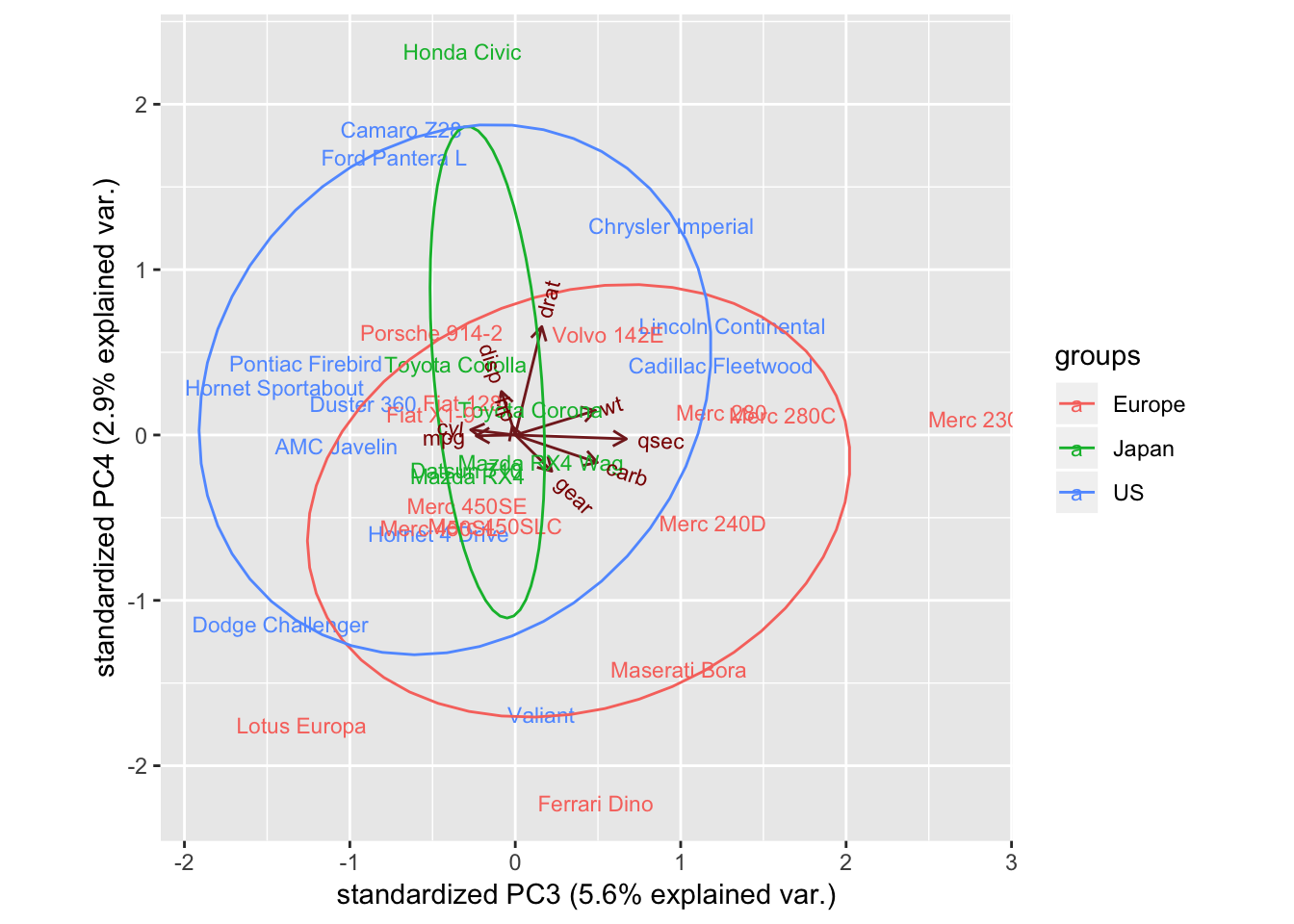
显然数据没有什么特点,这并不奇怪,因为PC3和PC4仅能表征数据很少的一部分特征。
综上,cyl,disp,wt,mpg可以用于区分美国和日本产汽车,如果需要构建一个汽车产地的模型,这些变量可能非常有用。
参考
- 主成分分析(PCA)及Demo最大方差解释和最小平方误差解释
- 如何通俗易懂地讲解什么是 PCA 主成分分析
- 主成分分析(PCA)模型
- 数据什么时候需要做中心化和标准化处理
- Principal Component Analysis in R
- PCA on correlation or covariance?
- Why do we need to normalize data before principal component analysis (PCA)?
- How does centering the data get rid of the intercept in regression and PCA?